Les généticiens peuvent maintenant créer des millions de nouveaux virus et bactéries au laboratoire en l'espace de quelques minutes. Et il n'y a aucune réglementation pour les empêcher d'être délibérément disséminés, comme le rapporte ici le Dr. Mae-Wan Ho
Le texte original en anglais et les références sont accessibles sur le web par : www.i-sis.org.uk/deathByDNAShuffling.php
Willem P.C. Stemmer de l'Institut de Recherche Affymax à Palo Alto, en Californie, Etats-Unis, a publié un article dans la revue américaine Proceedings of the National Academy Science en 1994, décrivant une technique de laboratoire dénommée ' Gene Shuffling ' et qui permet de procéder rapidement à une évolution moléculaire dirigée au moyen de recombinaisons aléatoires de l'ADN, avec des performances nettement améliorées.
Selon Stemmer, les études de simulation sur ordinateur avaient démontré l'importance de la recombinaison des segments des gènes dans l'évolution, plutôt que par de simples changements au niveau des bases. Pour mettre ceci en pratique, il a conçu une méthode qui permet de réassembler des gènes dans un tube à essais, à partir de fragments aléatoires de gènes.
L'ADN du gène est digérée avec l'enzyme désoxyribonucléase 1 (DNAse 1), aboutissant à des fragments aléatoires de 10 à 50 paires de base. Ces fragments sont chauffés jusqu'à leur séparation en deux brins puis ils sont ensuite réassociés en présence d'une ADN polymérase, une enzyme qui synthétisent l'ADN.
Les brins d'ADN séparés se réassocient selon des séquences de bases complémentaires avec de l'ADN (de l'ADN qui présente des séquences de bases similaires, en provenance de la même espèces ou d'espèces apparentées). Une séquence plus courte, associée avec une plus longue, va amorcer la synthèse en utilisant la plus longue séquence comme modèle, jusqu'à ce que le double brin d'ADN soit reconstitué. Avec des fragments réassociés de manière aléatoire, l'ADN polymérase peut mettre en contact des suites de bases de nombreuses fois au cours des réassociations et c'est ainsi que sont générés différentes formes de recombinaisons. Le diagramme simplifié ci-après ( Figure 1 ) indique ce qui est effectivement réalisé dans une expérience typique.
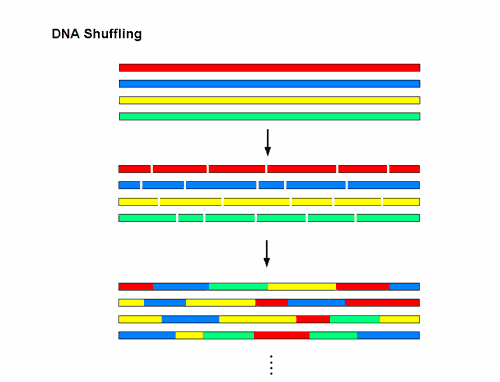
Figure 1 - ' ADN [ou gène] Shuffling '. Différentes variantes d'un gène ou d'une séquence d'ADN, représentées par les différentes couleurs, sont réduites en fragments et puis réassociées pour produire des millions de recombinants d'un seul coup.
Les recombinants sont clonés [c'est à dire copiés] dans un vecteur et introduits dans des bactéries en vue d'un criblage rapide.
De cette façon, beaucoup de variantes de n'importe quel gène peuvent être rapidement recombinées, même des variantes d'espèces différentes. De plus grands morceaux d'ADN, tels que des génomes entiers de plasmides , de virus et de bactéries peuvent également être recombinés. Pour augmenter la diversité de recombinants, de courtes séquences d'ADN synthétique peuvent être ajoutées dans le mélange. Même de l'ADN non homologue, provenant d'espèces n'ayant aucun lien de parenté, peut être recombiné.
Dans la toute première expérience rapportée, Stemmer avait prétendu avoir produit un recombinant d'une bêta-lactamase qui augmentait de 16.000 fois la résistance à l'antibiotique bêta-lactame. Quand ce recombinant était 'backcrossé' [croisé à nouveau en retour] avec le gène parental et en utilisant à nouveau la même technique de ' Gene Shuffling ' - qu'il appellera plus tard, 'multiplication moléculaire ' - une enzyme qui était 32.000 fois plus efficace que l'enzyme 'sauvage' [d'origine] avait été obtenue.
Précédemment, les chercheurs avaient employé d'autres méthodes pour introduire des erreurs aléatoires, telles que la réaction en chaîne de polymérase (PCR), mais cela n'avait donné, dans le meilleur des cas, qu'une amélioration de 16 fois. Et la 'conception rationnelle', basée sur la structure connue de l'enzyme n'avait pas fait mieux. Ainsi les résultats obtenus avec ce ' Gene Shuffling ' semblent hors du commun.
Stemmer a monté depuis cette époque une société basée en Californie, Maxygen Inc. pour exploiter cette technologie. Dans une série de publications entre 1998 et 2002, lui et ses collaborateurs au sein de Maxygen Inc. , ainsi que d'autres sociétés, ont employé la technique pour obtenir les protéines considérablement améliorées, telles que la protéine verte fluorescente, le fucosidase, la subtilisine, la thymidine kinase et l'interféron-alpha fluorescent vert, de même que des vecteurs rétroviraux améliorés et des souches bactériennes de Streptomyces et de Lactobacillus .
Dans l'expérience sur des génomes rétroviraux, six virus de la leucémie des souris (MLV) ont été recombinés dans une simple structure circulaire, donnant un assemblage de cinq millions de virus recombinés et capables de se répliquer. Parmi eux se trouvaient des virus complètement nouveaux et qui pouvaient infecter les cellules d'ovaire du hamster de Chine, qu'aucun variant du virus MLV original n'était capable d'infecter, ainsi que des virus recombinés qui étaient 30 à 100 fois plus stables que les souches parentales, et par conséquent bien mieux adaptés pour la thérapie génique.
Jusqu'ici, de telles expériences semblent avoir été effectuées exclusivement par Maxygen Inc . ou par cette société en collaboration avec d'autres, et donc n'avoir été pas répétées indépendamment, en ce qui concerne ces remarquables succès.
Il n'y a aucun doute, cependant, que le procédé est dangereux en soi. Comme ces chercheurs l'ont amplement démontré, la recombinaison est le moyen principal pour produire de nouveaux virus et bactéries. Certains de ces virus et bactéries recombinés pourraient tout aussi bien être mortels.
Quelles précautions ont été prises en ce qui concerne les recombinants et les vecteurs employés, pour les empêcher d'être disséminés dans l'environnement ? Les virus et les bactéries recombinées, ou leur ADN, ont-il été strictement confinés dans le laboratoire?
La réglementation en vigueur sur l'utilisation confinée des microorganismes génétiquement modifiés est tout à fait inadéquate, et nous avons à plusieurs reprises signalé ceci aux autorités chargées de cette réglementation. En Europe, des ' disséminations tolérées ' sont autorisées , et ceci inclut non seulement les déchets transgéniques se composant de bactéries et de cellules mortes (avec plein d'ADN transgénique), mais également certaines bactéries vivantes considérées, par les sociétés concernées, comme ne constituant pas une menace pour la santé ou pour l'environnement.
Avec des expériences non réglementées, comme cela se passe dans ces sociétés, avons-nous encore besoin de bioterroristes ?
Article first published 17/04/03
Définitions et compléments d'information en français :
' Gene ou ADN Shuffling ' ou évolution moléculaire dirigée par recombinaisons aléatoires : les technologies d'ingénieurie des protéines, notamment enzymatiques, tendent à imiter, par accélération des processus, l'évolution naturelle et à produire ainsi de nouvelles molécules. Lors de la mise en œuvre des techniques de PCR, on est en mesure d'obtenir des dizaines de milliers de nouvelles combinaisons génétiques au bout de quelques heures. Ces combinaisons permettent alors de sélectionner des protéines présentant des propriétés modifiées ou des fonctions biochimiques entièrement nouvelles. Pour illustrer ces applications, l'on peut se reporter à une é tude en français sur l'ingénieurie des enzymes : évolution dirigée , randomisation de l'ADN, recombinaison aléatoire par « gene shuffling », avec des exemples expérimentaux, en consultant le site suivant : mapageweb.umontreal.ca/keillorj/ Notes/Evolution%20dirigee.pdf
Plasmide : c'est une petite portion circulaire d'ADN extra chromosomique (beaucoup plus petit que le chromosome) que l'on retrouve principalement dans le cytoplasme des bactéries, où il s'y multiplie de manière autonome, de façon indépendante. Ce fragment d'ADN est stable et autoreproducteur. Le plasmide contient des caractères génétiques non essentiels à la cellule bactérienne hôte, mais également certains gènes importants pour la bactérie hôte, tels que des gènes de résistance à des antibiotiques ou des gènes de synthèse de certaines molécules. Les plasmides peuvent être transmis d'une cellule mère vers ses cellules filles (transmission verticale) ou entre bactéries (transmission génétique horizontale). Les plasmides constituent le principal véhicule d'insertion d'informations génétiques nouvelles, ou vecteur de clonage, dans les microorganismes ou chez les végétaux, lors du processus de transgénèse : il est possible de cloner des fragments d'ADN d'une taille de 50 à 50.000 paires de base.
Traduction, définitions et compléments en français :
Jacques Hallard, Ing.CNAM, consultant indépendant
Adresse : 19 Chemin du Malpas 13940 Mollégès France
Courriel : jacques.hallard@wanadoo.fr
Comments are now closed for this article